

当单细胞测序的UMAP图上出现一个孤零零的cluster游离在“上皮-免疫” 细胞群之外——它没有常规Marker基因的“身份证明”,在数据库里也查无此人。这不是生信分析的终点,而是一场“细胞身份解码”的开始。在单细胞研究中,聚类得到的cluster0、cluster1……只是一串冰冷的编号,而细胞注释,正是把这些编号翻译成“T细胞”“癌细胞”“巨噬细胞”的“翻译器”,堪称单细胞测序的“第二大脑”。单细胞测序让我们能看清组织中每一个细胞的基因表达,但真正的挑战在于:如何给这些细胞“上户口”?
一、细胞注释的核心逻辑:从 “数字编号” 到 “生物身份”
细胞注释的本质,是通过基因表达特征 “反向推导” 细胞的功能身份。下图中提到的注释流程(图1)清晰展现了这一过程,简单来说分为三步:自动注释打基础、手动注释辨细节、多方验证保准确。
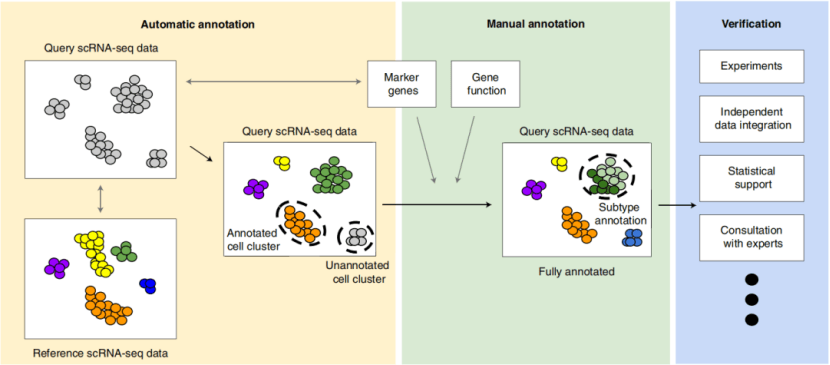 图1 细胞注释流程[1]
图1 细胞注释流程[1]
(1) 两个层面的注释:既认“细胞身份”,也读“基因密码”
细胞注释不是单一维度的工作,而是从两个层面展开:
举个例子,当某个cluster高表达Col3al基因,结合GO注释发现它富集在“胶原合成”通路,我们就能推测这可能是成纤维细胞——既认对了“细胞身份”,也读懂了它的“功能密码”。
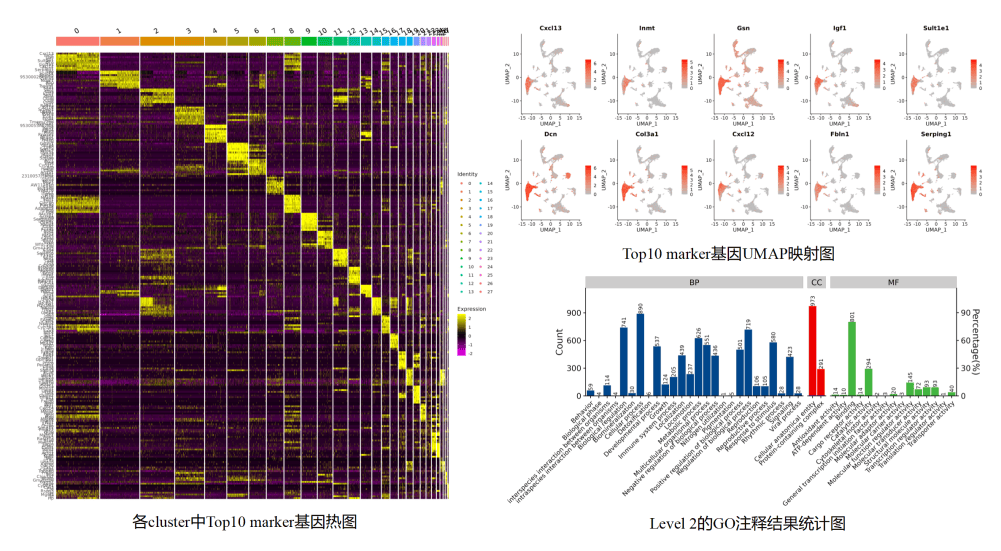 图2 细胞Marker基因可视化
图2 细胞Marker基因可视化
(2) 三步搞定细胞注释:从工具到逻辑
想要给细胞“精准上户口”,以下总结的“三步法”堪称实用指南:
第一步:参考数据集 “自动匹配”
就像用“人脸比对”找熟人,把待分析数据与公共参考数据集(如CellMarker2.0、PangoDB)比对。比如CellMarker2.0收录了人类445万+细胞的Marker基因,输入基因名称就能查到它常见于哪种细胞——这一步能快速锁定“嫌疑细胞类型”图3)。
第二步:经典 Marker 基因 “贴标签”
如果自动匹配有歧义,就用 “典型Marker” 来验证。比如上皮细胞高表达EPCAM,免疫细胞高表达CD45,这些经过大量研究验证的“身份标签”,能帮我们排除错误注释。Seurat、Scanpy等工具就是通过这类基因,给细胞群 “盖戳认证”。
第三步:功能特征 “深度验身”
对“身份可疑”的细胞,得看它的“行为特征”:通过差异基因分析找到它独有的高表达基因,再用GO、KEGG富集分析看这些基因参与哪些通路(比如“细胞增殖”“免疫应答”),甚至用GSVA分析它的通路活性。这一步能帮我们发现“新亚型”——比如同样是巨噬细胞,有的可能高表达炎症基因,有的则偏向血管生成。
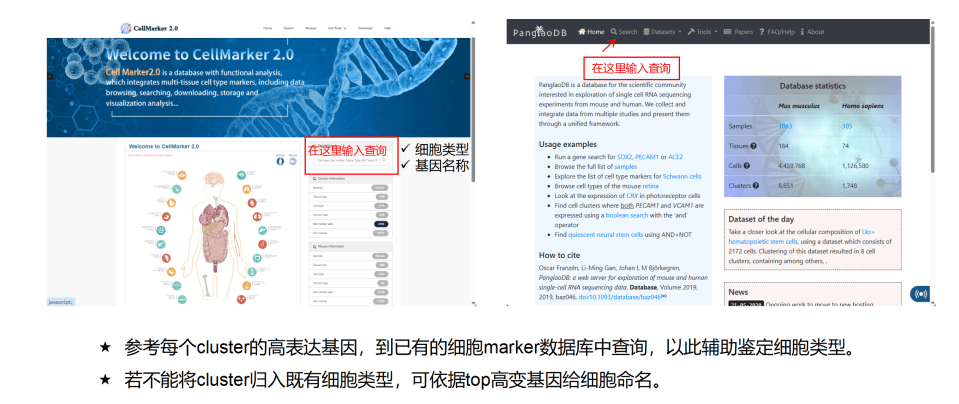 图3 细胞Marker基因查询网站
图3 细胞Marker基因查询网站
二、肿瘤中的细胞注释:不只是“认人”,更要“抓坏人”
肿瘤组织的细胞注释,堪称“最复杂的身份识别现场”。这里不只是癌细胞的“独角戏”,还有免疫细胞、成纤维细胞、内皮细胞等“群演”,甚至癌细胞自身还会“伪装”“变异”,给注释带来大挑战。
(1) 肿瘤样本的单细胞数据复杂在于:
因此,仅靠传统marker难以胜任,需要基因表达特征+CNV异常双重判断。
(2) 癌细胞表达与差异
▶使用癌症相关marker库看表达:如TCGA、OncoKB中已知的癌基因表达特征EPCAM、KRTs(上皮性癌),CDKN2A、MKI67(增殖)等;
▶识别肿瘤 vs.非肿瘤细胞差异:结合CNV(inferCNV(图4)、CopyKAT)识别是否发生染色体拷贝数变异,区分恶性与非恶性细胞,CNV可辅助判断哪些细胞是肿瘤源。
(3) 功能状态注释
▶用GSVA分析肿瘤通路(如EMT、p53、hypoxia);
▶利用TISCH等数据库查看肿瘤类型下的免疫细胞参考表达谱。
 图4 inferCNV分析结果示例
图4 inferCNV分析结果示例
三、巨噬细胞注释:从“一把钥匙”到“多齿密码”
在细胞注释中,巨噬细胞是最“善变”的例子之一。传统上用CD68标记巨噬细胞,但就像一把多齿钥匙,每个“齿纹”都对应不同亚型:
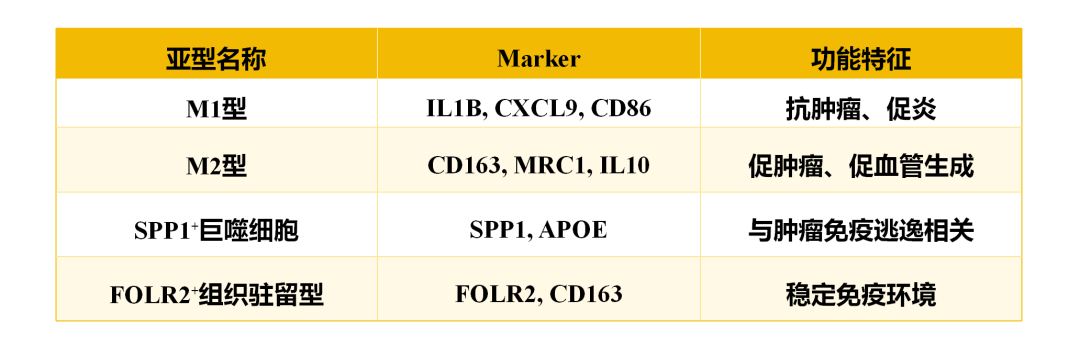
 图5 人类免疫细胞与亚型的marker注释示例[2]
图5 人类免疫细胞与亚型的marker注释示例[2]
四、遇到“查无此人”的细胞?5步攻略破解
当cluster既无Marker,又不匹配参考数据集,别慌!本文给出了“5步注释攻略”:
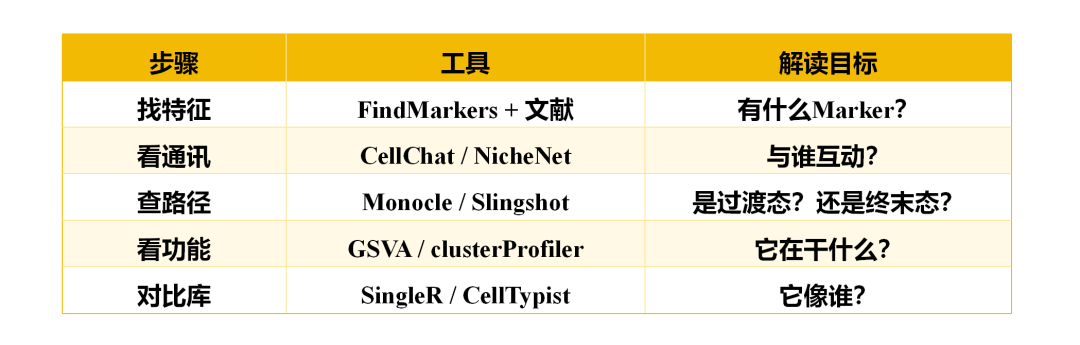
如果还是难确定,结合空间转录组看它的“位置”——比如靠近血管的细胞可能参与营养运输,靠近肿瘤的可能参与免疫抑制,位置信息能给注释“加buff”。
拜谱小结
单细胞测序为我们打开了“看清细胞”的大门,但走进细胞世界,离不开注释这一“语言翻译器”。从肿瘤微环境的混战中找到关键调控细胞,从免疫细胞的变化中发现疾病机制。拜谱生物依托10x Genomics、墨卓等单细胞平台,结合蛋白组、代谢组等多组学数据,配备丰富的marker数据库和标准化注释流程,帮你打通从“数据挖掘”到“生物学洞察”的最后一公里。欢迎咨询合作!
参考文献:
[1]Clarke ZA, Andrews TS, Atif J, et al. Tutorial: guidelines for annotating single-cell transcriptomic maps using automated and manual methods. Nat Protoc. 2021;16(6):2749-2764. doi:10.1038/s41596-021-00534-0
[2]Kock KH, Tan LM, Han KY, et al. Asian diversity in human immune cells. Cell. 2025;188(8):2288-2306.e24. doi:10.1016/j.cell.2025.02.017