


T细胞可以识别恶性细胞表面的主要组织相容性复合体(MHCs)上的肽表位,引起抗肿瘤免疫反应。理论上,抗肿瘤表位可能来自两类抗原:
第一类抗原称为肿瘤相关抗原(TAAs),它们在恶性肿瘤中异常表达或仅在分化的特定阶段产生,而在正常组织中的表达极为有限。TAAs是非突变的自身抗原,其临床应用受限于中央T细胞的耐受性。
第二类则是肿瘤细胞基因组突变(如SNVs、INDEL、基因融合、SVs)所产生的独特肽段,也被称为新生抗原或肿瘤特异性抗原(TSAs)。此外,新抗原也可能来源于异常转录组变异(如转录本选择性剪接、顺式作用元件突变、剪接因子反式作用的改变、无义介导的RNA衰减、多聚腺苷酸化和RNA编辑、非编码区)、蛋白质组变异(如翻译后修饰(PTMs)异常、蛋白酶体加工受损或TAP复合物)和病毒ORF等(图1)。与TAAs相比,新抗原在其独特的肿瘤特异性方面具有明显的优势(表1),将为有效的肿瘤个性化治疗提供理想的靶点。
表1 TAAs和TSAs的特点
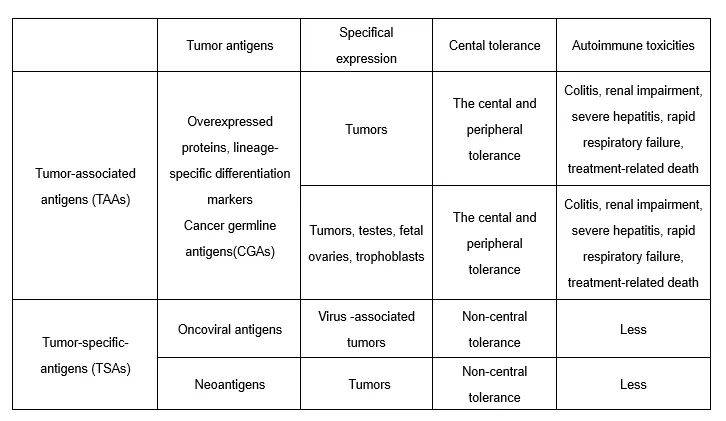 (表源. Xie et al.Signal Transduction Targeted Ther.2023)
(表源. Xie et al.Signal Transduction Targeted Ther.2023)
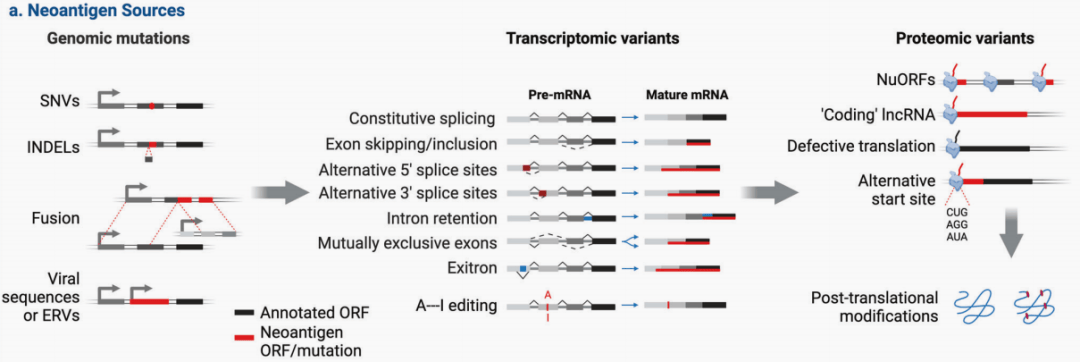 图1 肿瘤新生抗原的产生
图1 肿瘤新生抗原的产生
(图源:Xie et al.Signal Transduction Targeted Ther. 2023)
01、免疫原性新抗原的鉴定
免疫基因组学策略是利用NGS来比较肿瘤和正常组织之间的遗传变化。目前,从NGS数据中检测可能新抗原是利用肿瘤和正常的RNA/DNA测序数据来定位肿瘤特异性基因异常。同时,RNA-seq数据与WES结合可以确定肿瘤组织中表达的突变基因,RNA-seq还可以发现更多隐藏的生物信息,如拷贝数变化、微生物污染、转座因子、细胞类型等信息。RNA-seq也可用于检测选择性剪接事件,并估计突变等位基因表达的相对频率。最近的研究表明,当假定无义衰变(NMD)存在时,抗原多肽是由具有移码突变和非典型剪接模式的转录本产生的,为了完全识别由移码突变和异常亚型导致的新抗原,需要来自全长转录本结构的精确肽序列。
基于质谱技术的免疫肽组学可以直接检测免疫沉淀提取的MHC结合多肽,实现高通量鉴定MHC上呈递的多肽(图2)。除了由异常的DNA序列或RNA表达引起的新抗原外,基于MS的蛋白质组学还为蛋白质水平上的新抗原检测提供了“金标准”,这是从DNA和RNA研究中无法发现的。例如,MS可用于检测由细胞转化过程中PTMs失调引起的新型MHC相关新抗原。此外,MS与NGS集成,可进一步检测体细胞突变、非编码RNA和蛋白酶体剪接产生的肿瘤特异性新抗原。整合基因组、转录组和蛋白质组数据,将有利于基于免疫肽组的新抗原鉴定。
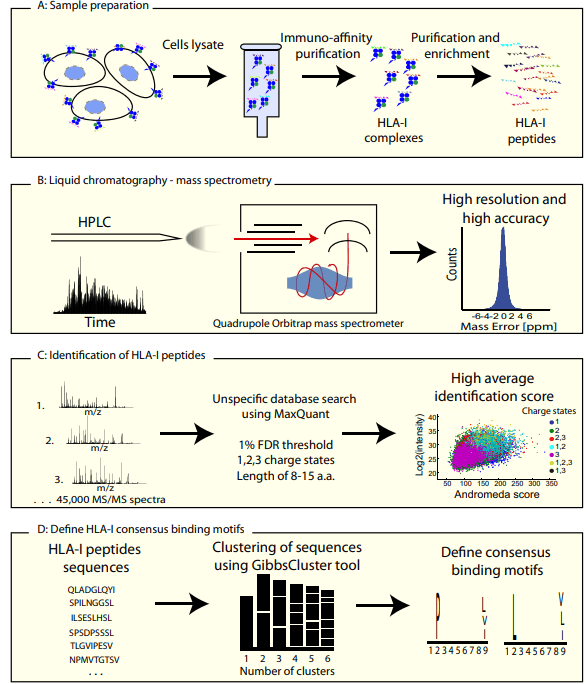 图2 基于质谱的新抗原鉴定技术路线
图2 基于质谱的新抗原鉴定技术路线
(图源. Bassani-Sternberg, Michal et al.MCP. 2015 )
02、计算机辅助预测新抗原
新抗原预测的典型工作流程可以总结为以下步骤:(i)突变识别,(ii)HLA分型,(iii)基于HLA结合亲和力的新抗原过滤和优先排序,以及(iv)使用基于T细胞的检测对免疫原性新抗原进行实验验证(图3)。
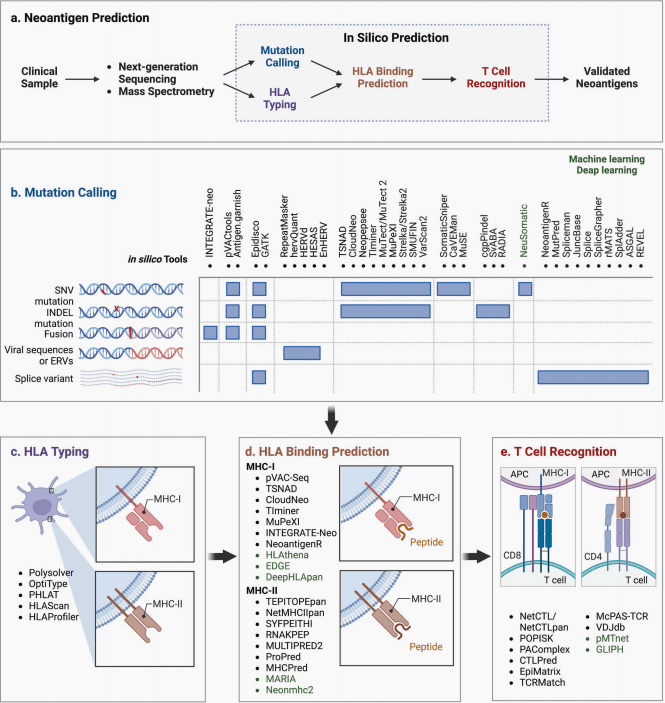 图3 新抗原预测的计算工作流程
图3 新抗原预测的计算工作流程
(图源:Xie et al.Signal Transduction Targeted Ther. 2023)
基于MHC分子的加工和呈递过程,已经创建了许多用于计算机预测的工具来发现新抗原,包括NetChop、NetCTL和NetCTLpan等(图3)。通过将HLA-配体数据纳入机器学习算法,已经显著提高了预测能力。NetMHCpan和MHCflurry利用体外Peptides-HLA Binding数据集来训练机器学习模型,是当前HLA配体识别管道的主要组成部分,与最先进的方法相比,NetMHCpan通过整合结合亲和力数据和MS多肽组数据,为MHC-I等位基因提供一种“全特异性”机器学习策略,提高了肿瘤新抗原的预测性能。最近的两项研究创建了名为MSIntrinice和EDGE的计算框架,利用从RNA-seq和液相色谱串联质谱(LC-MS/MS)数据中获得的HLA肽,可以高效地预测HLA抗原。
新的证据已经证明了MHC-II新抗原在抗肿瘤免疫反应中的重要意义。利用人工神经网络开发了大量预测MHC-II结合表位的计算技术,包括NetMHCII、NetMHCIIpan、RNAKPEP、MULTIPRED2、ProPred和MHCPred。然而,与MHC-I分子相比,MHC-II-peptides结合亲和力的计算预测目前还不那么精确。近年来,基于转录组和质谱数据的计算方法已经发展起来。在淋巴瘤数据训练集中,由MARIA训练的深度学习模型优于最广泛使用的NetMHCIIpan3.1。然而,还需要更多的研究使用重要的数据集来证明其稳健性和有效性。
大多数通过MHC分子呈递预测的新抗原不会触发免疫反应。因此,在评估潜在新抗原的免疫原性时,尤其需要考虑pMHC复合物的TCR识别。计算机模型可以预测新抗原特异性T细胞的识别,如NetCTL/NetCTLpan和基于机器学习的TCR-peptides/-pMHC binding。然而,由于TCR对其pMHC配体的低亲和力,预测TCR和pMHC的结合亲和力仍然具有挑战性。
03、候选新抗原免疫原性的评价与验证
新抗原反应性T细胞通常利用T细胞检测、多色标记的MHC四聚体、酶联免疫吸附点(ELISpot)和T细胞文库分析进行验证或筛选。T细胞免疫原性检测是评价候选新抗原免疫原性最直接的方法。通过癌症外显子组/RNA-seq发现的一整套可能的突变多肽,都可以使用来自癌症患者或健康捐赠者的T细胞进行检测。这些技术依赖于表位预测,通量较低,因为它们只能有效地生成MHC I类等位基因的一个子集。将单细胞RNA测序(scRNA-seq)与应答细胞群的TCR测序相结合,可用于提高检测的灵敏度。
一些无偏的TCR-guided新抗原发现策略已经被开发出来系统地描述新抗原特异性TCR。酵母展示的pMHC文库可用于发现新抗原特异性TCR,然而,制备可溶性TCR试剂是一个耗时的过程,如果没有新抗原的内源性处理或T细胞的功能激活,所识别的随机肽可能不能代表生理上的TCR-pMHC相互作用。为了克服这些缺点,可以利用不同的生物过程在共培养系统中标记目标细胞。一种方法利用被称为信号传导和抗原呈递双功能受体(SABRs)的嵌合受体,它可以在pMHC-TCR相互作用后诱导TCR样信号。SABRs能够成功识别TCR-pMHC相互作用,可用于已知的公共TCR和个体新抗原特异性TCR。T-Scan是一种独立于预测算法的TCR表位扫描方法,它依赖于T细胞杀伤的生理活性,比以前的方法产生更大的抗原空间。因此,这些发现TCR配体的新兴方法将有助于研究候选新抗原的免疫原性,为免疫治疗提供新的靶点。
04、总结
下一代测序技术和生物信息学技术的不断发展,加速了肿瘤特异性新抗原的快速识别和预测。通过了解新抗原诱导的抗肿瘤免疫反应的机制,以及通过简化基于新抗原的免疫治疗的过程,将有助于癌症治疗的快速发展。
拜谱小结
肿瘤新生抗原(neoantigen)是由体细胞突变产生的能诱导肿瘤特异性T细胞识别的多肽,是一种肿瘤特异性抗原,是肿瘤免疫治疗的理想靶点。目前基于新生抗原的研发已被广泛应用于肿瘤疫苗、过继性免疫细胞治疗、抗体药等多个方面。新抗原的鉴定是开发有效免疫疗法的关键步骤。鉴定新抗原表位有两种主要策略:①免疫基因组学方法:基于高通量测序的计算机方法创建虚拟肽组;②免疫肽组学方法:使用质谱分析MHC结合肽段。拜谱生物作为国内领先的多组学公司,可提供完善成熟的蛋白组学、代谢组学、转录组学等多组学产品技术服务体系。近期,拜谱生物将推出基于质谱分析的免疫肽组检测服务,敬请期待!
参考文献
1.Xie N,Shen G,Gao W, et al. Neoantigens: promising targets for cancer therapy. Signal Transduct Target Ther. 2023;8 (1):9. doi:10.1038/s41392-022-01270-x
2.Bassani-Sternberg M,Pletscher-Frankild S,Jensen LJ, et al. Mass spectrometry of human leukocyte antigen class I peptidomes reveals strong effects of protein abundance and turnover on antigen presentation. Mol Cell Proteomics. 2015;14 (3):658-73. doi:10.1074/mcp.M114.04281