CHIP(Chromatin Immunoprecipitation,染色质免疫沉淀)技术是普遍使用的研究蛋白质和DNA互作的应用。CHIP-seq将染色质免疫沉淀与高通量测序技术结合,可以鉴定蛋白质在全基因组上的结合位点。细胞染色质由 DNA 和组蛋白组成。组蛋白可以进行多种翻译后修饰(Post translational modification,PTM),例如磷酸化、甲基化、乙酰化和其他酰化修,也就是表观遗传修饰;DNA可以通过甲基化修饰以调节基因表达。CHIP-seq可以作为研究组蛋白表观遗传修饰和转录因子结合基因进行转录调控的利器。
实验过程
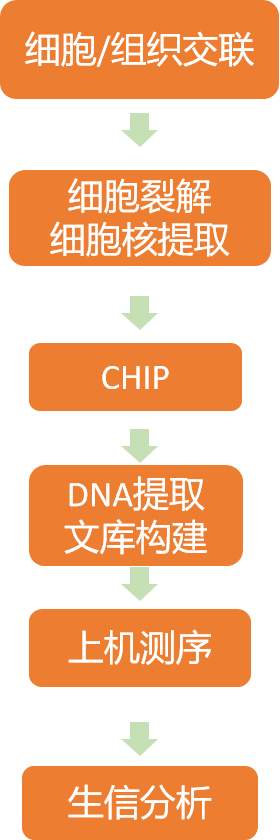
拜谱生物提供服务:
类型 | 数据量 | 客户提供 | 拜谱提供 |
CHIP-seq | 10M reads | 目标蛋白信息 样品和抗体 | 免疫共沉淀 建库测序 生物信息学分析 |
CHIP完成后,做建库测序 | 10M reads | 目标蛋白信息 样品 | 建库测序 生物信息学分析 |
样本要求:
细胞培养:>2*10⁶个;动物组织:>500mg;植物组织:>1g;
参考文献:
Orlando DA, Chen MW, Brown VE, et al. Quantitative ChIP-Seq normalization reveals global modulation of the epigenome. Cell Rep. 2014;9(3):1163-1170
拜谱生物收样表下载: 基因组学送样表(拜谱生物).xls
基因组学送样表(拜谱生物).xls